近日,我院毛胜勇教授团队联合吉林农业大学动物科学技术学院李志鹏教授团队、西北工业大学生态与环境学院邱强教授团队以及中科院亚热带农业生态研究所王敏研究员团队,完成了反刍动物胃肠道微生物组基因和基因组集的构建。相关文章在微生物学权威期刊《Microbiome》发表:An integrated gene catalog and over 10,000 metagenome-assembled genomes from the gastrointestinal microbiome of ruminants。
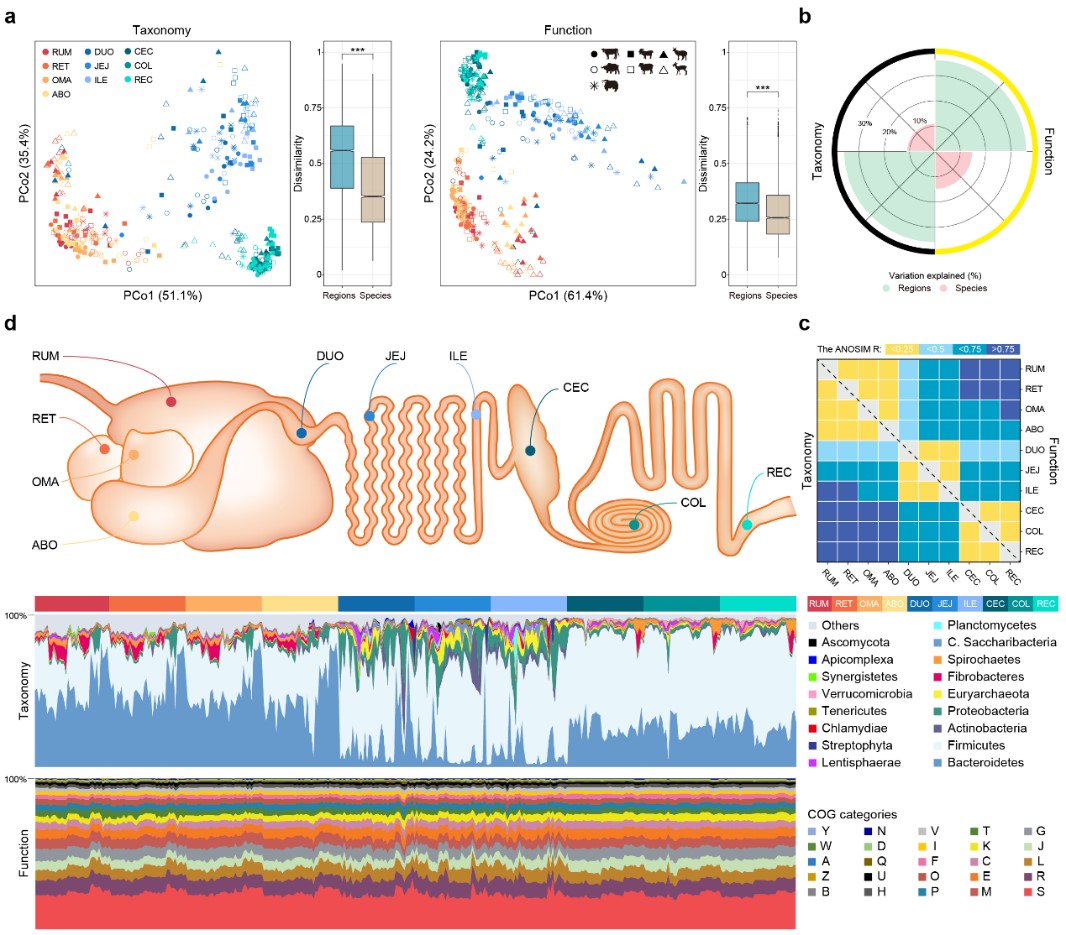
反刍动物能够利用植物纤维物质生产肉和奶等高营养价值的食品,其主要取决于反刍动物消化道微生物群落的特殊结构与功能。长期以来,人们重点研究了反刍动物瘤胃微生物的菌群组成和功能,但近年来研究人员发现,反刍动物小肠和后肠微生物对饲料效率和宿主健康也具有重要作用。为阐明反刍动物全胃肠道微生物组的结构和功能,明晰其与宿主健康及饲料效率间的内在关联。项目选取了7种代表性反刍动物,包括奶牛、水牛、牦牛、山羊、绵羊、狍子和獐子,分别采集了这些动物全胃肠道10个区段的食糜样品,对370个样品进行了宏基因组测序。项目首次构建了反刍动物胃肠道微生物基因集(非冗余基因>154 M),该基因集为反刍动物胃肠道微生物组的研究提供了参考,同时为跨物种和多领域的微生物组比较研究提供了数据支撑。项目同时组装了超过10,000个非冗余的微生物基因组,在种水平上鉴定出了8,745个新基因组,这些基因组极大的拓展了反刍动物消化道微生物基因组资源,在分类学上大幅提升了对反刍动物消化道微生物的分辨率,这些新发现的微生物资源对反刍动物消化道微生物的分离培养、甲烷减排和高效生产的研究提供了新的支撑。
南京农业大学博士生谢斐、金巍副教授、吉林农业大学司华哲博士和西北工业大学博士生袁源为文章的共同第一作者。该研究得到了国家自然科学基金和南京农业大学基础前沿研究等项目的支持。